搜索


近日,英国抗菌化疗协会 (British Society for Antimicrobial Chemotherapy)出版的刊物《Journal of Antimicrobial Chemotherapy》在线发表了上海交通大学微生物代谢国家重点实验室邓子新团队关于肺炎克雷伯菌HS11286的基因组分析论文“Mapping the resistance-associated mobilome of a carbapenem-resistant Klebsiella pneumoniae strain reveals insights into factors shaping these regions and facilitates generation of a ‘resistance-disarmed’ model organism” 。该论文的第一作者为博士生毕德玺,指导教师为欧竑宇教授。
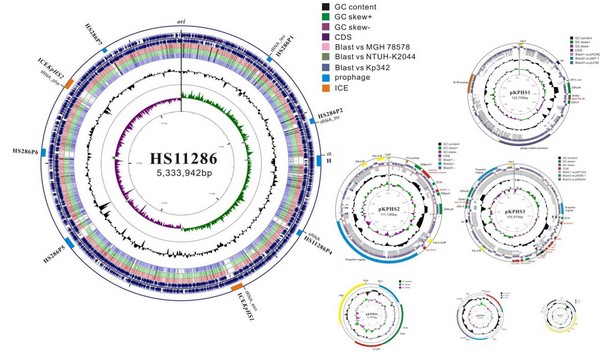
抗生素滥用引发的细菌耐药问题已成为全球关注热点之一,应对以耐碳青霉烯类抗生素为代表的多重耐药菌已刻不容缓;而肺炎克雷伯菌 (Klebsiella pneumonia) 则是院内感染最重要的多重耐药条件致病菌之一。该研究深入分析了碳青霉烯类抗生素抗性的、属于中国流行克隆群 (ST11型) 的肺炎克雷伯菌HS11286基因组,发现了6个质粒、2个整合性接合元件和7个前噬菌体等复杂的可移动遗传元件,阐明了转座子Tn3、Tn1721和Tn5393和插入序列IS26等元件对blaKPC-2等二十多个重要耐药基因的水平转移有重要贡献。此外,辅于分子遗传学实验,确定了HS11286的耐药基因缺失突变株对临床常用抗菌药物的敏感性大幅提高,由此也成功构建了可用于实验室安全遗传操作的肺炎克雷伯菌模式菌株。
这是该团队在病原细菌基因组研究中取得的最新进展之一。此外,今年他们还合作报道了转座子Tn1721同时编码碳青霉烯酶和磷霉素抗性 (Antimicrobial Agents and Chemotherapy, 2015, 59:338-343),比较分析了高致病鲍曼不动杆菌的全基因组序列 (Scientific Reports, 2015, 5:8643)。这些进展得益于该团队围绕细菌可移动基因组研发的微生物信息学平台。近十年来,该平台向国内外微生物学同行提供了细菌基因组快速比对和可移动遗传元件识别等一系列特色的在线工具和开放数据库。其中,与细菌耐药性播散相关的整合性接合元件数据库ICEberg和IV型分泌系统数据库SecReT4、与细菌致病性相关的VI型分泌系统数据库SecReT6等三个分子生物学数据库被美国橡树岭国家实验室的Dr. Igor B. Zhulin向微生物学研究者推荐使用 (J Bacteriol, 2015, 197:2458-67)。这些研究是微生物代谢国家重点实验室在分子微生物学和生物信息学等多学科交叉所取得的一系列进展,将有助于实现重要微生物代谢潜力的深度挖掘。


 英国上市公司官网365
英国上市公司官网365

 学院概况
学院概况

 师资队伍
师资队伍

 人才培养
人才培养

 招生就业
招生就业

 科学研究
科学研究

 平台基地
平台基地

 党群工作
党群工作

 校友之家
校友之家

 安全工作
安全工作

 网上办事
网上办事

 当前位置:
当前位置: